TIANSeq komplet RNA-Seq komplet (ilumina)
Značajke
■ Dobra ujednačenost sekvenciranja: Visoka vjernost PCR amplifikacije i bez pristranosti baze.
■ Visoka učinkovitost pretvorbe knjižnice: Visokoučinkovita konstrukcija knjižnice može se osigurati za uzorke mRNA veličine 1 ng.
■ Brz rad: Cijelom procesu izgradnje knjižnice potrebno je samo 5,5 sati.
Specifikacija
Tip: Priprema NGS biblioteke usmjerene RNK
Uzorak: Ukupna RNK
Cilj: mRNA, lncRNA
Početni unos uzorka: Ukupni uzorci RNA su 10 ng-1 μg, a uzorak mRNA je nizak kao 1 ng
Vrijeme rada: 5,5-6,5 sati
Nizvodne aplikacije: Sekvenciranje na platformi illumina.
Svi se proizvodi mogu prilagoditi za ODM/OEM. Za detalje,kliknite Prilagođena usluga (ODM/OEM)
Usporedba knjižničnog prinosa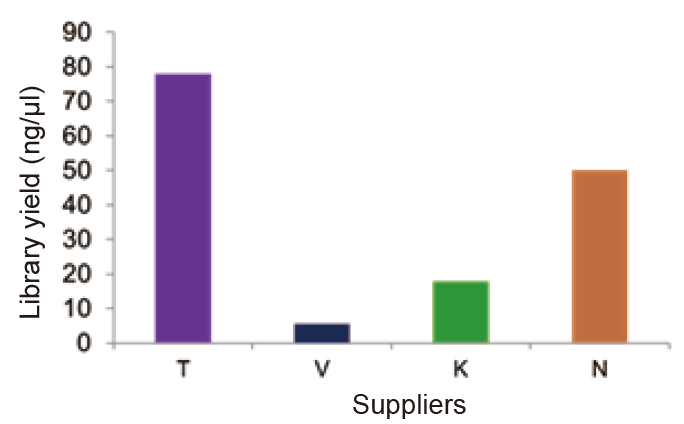 |
Slika 1. Knjižnica RNA od 1 μg ukupne RNK humane 293T stanice izgrađena je pomoću TIANSeq Stranded RNASeq Kit i relevantnih proizvoda iz Dobavljača V, K i N. Rezultat pokazuje da TIANSeq Stranded RNA-Seq Kit ima značajno veći prinos od onog iz Dobavljača V , K i N. |
Ujednačena pokrivenost transkriptom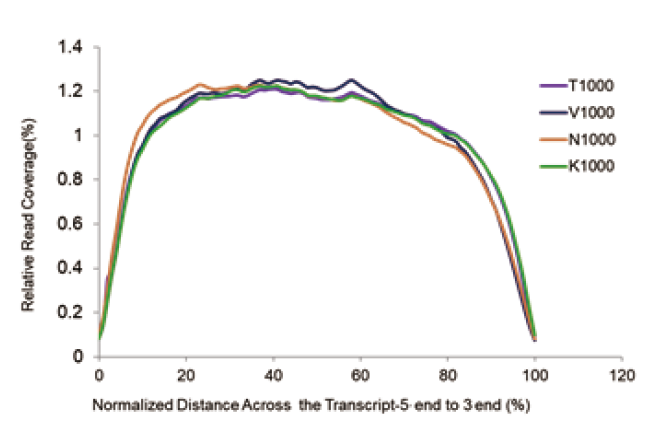 |
Slika 2. RNK biblioteka od 1 μg ukupne RNA humane 293T stanice izgrađena je korištenjem TIANSeq Stranded RNASeq Kit -a i relevantnih proizvoda dobavljača V, K i N. Rezultat pokazuje da je TIANSeq Stranded RNASeq Kit imao vrlo jednoličnu pokrivenost od 5 'do 3' kraj, koji je bio bolji od dobavljača V, a performanse su bile slične proizvodima dobavljača N i K. |
Širok raspon primjene ulaznog uzorka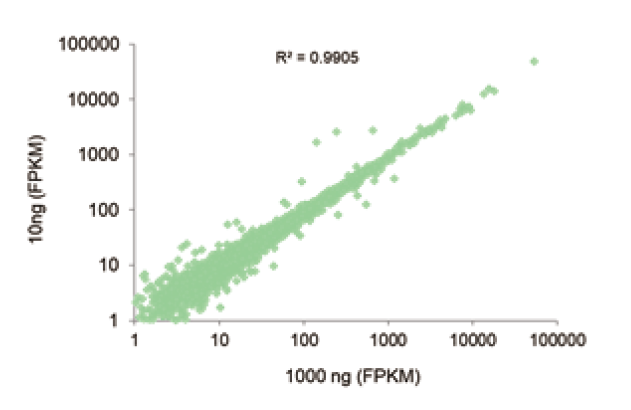 |
Slika 3. TIANSeq Stranded RNA-Seq Kit je kompatibilan s 10 ng-1 μg ukupnog unosa RNA, a biblioteka izgrađena pod različitim ulazima uzorka ima visoku konzistenciju. Rezultat pokazuje da je koeficijent korelacije ekspresije gena između knjižnica dosegao iznad 0,99 pod ukupnim unosom RNA od 10 ng i 1 μg stanice 293T, a konzistentnost podataka bila je visoka. |
Trenutno se visokopropusna tehnologija sekvenciranja uglavnom temelji na tehnologiji sekvenciranja sljedeće generacije. Kako je duljina čitanja sljedeće generacije tehnologije sekvenciranja ograničena, moramo rastaviti niz cijele duljine u male biblioteke fragmenata za slijed. U skladu s potrebama različitih eksperimenata sekvenciranja, obično odabiremo jednostruko ili dvosmjerno sekvenciranje. Trenutno se fragmenti DNA biblioteke za sekvenciranje sljedeće generacije općenito distribuiraju u rasponu od 200-800 bp.

a) DNK je loše kvalitete i sadrži inhibitore. Koristite visokokvalitetne uzorke DNK kako biste izbjegli inhibiciju aktivnosti enzima.
b) Količina uzorka DNA nije dovoljna kada se za izradu DNK biblioteke koristi metoda bez PCR-a. Kad unos fragmentirane DNA pređe 50 ng, tijek rada bez PCR-a može se selektivno provesti tijekom procesa izgradnje knjižnice. Ako je broj kopije biblioteke prenizak da bi se izravno sekvencirao, DNK biblioteka se može amplificirati PCR -om nakon ligacije adaptera.
c) Kontaminacija RNA dovodi do netočne početne kvantifikacije DNA Kontaminacija RNA može postojati u procesu pročišćavanja genomske DNA, što može dovesti do netočne kvantifikacije DNA i nedovoljnog učitavanja DNA tijekom izgradnje knjižnice. RNA se može ukloniti tretiranjem s RNazom.
A-1
a) Pojavljuju se mali fragmenti (60 bp-120 bp) Mali fragmenti su obično fragmenti adaptera ili dimeri nastali pomoću adaptera. Pročišćavanje pomoću magnetskih kuglica Agencourt AMPure XP može učinkovito ukloniti ove fragmente adaptera i osigurati kvalitetu sekvenciranja.
b) Veliki se fragmenti pojavljuju u knjižnici nakon PCR amplifikacije. Veličina knjižničnog fragmenta DNA povećat će se za 120 bp nakon što se adapter poveže. Ako se fragment DNA poveća za više od 120 bp nakon ligacije adaptera, to može biti uzrokovano abnormalnom amplifikacijom fragmenta prekomjernom amplifikacijom PCR -a. Smanjenje broja PCR ciklusa može spriječiti situaciju.
c) Nenormalna veličina DNK fragmenata biblioteke nakon ligacije adaptera Duljina adaptera u ovom kompletu je 60 bp. Kad se dva kraja fragmenta povežu s adapterima, duljina će se povećati samo za 120 bp. Kad koristite adapter koji nije priložen u ovom kompletu, obratite se dobavljaču radi pružanja relevantnih informacija, poput duljine adaptera. Molimo provjerite slijede li tijek rada i rad eksperimenta korake opisane u priručniku.
d) Nenormalna veličina fragmenta DNA prije ligacije adaptera Razlog ovog problema može biti uzrokovan pogrešnim reakcijskim uvjetima tijekom fragmentacije DNA. Za različit unos DNK treba koristiti različita vremena reakcije. Ako je unos DNA veći od 10 ng, preporučujemo da za početak optimizacije odaberete vrijeme reakcije od 12 minuta, a veličina fragmenta koji se tada proizvodi uglavnom je u rasponu od 300-500 bp. Korisnici mogu povećati ili smanjiti duljinu fragmenata DNA za 2-4 minute prema vlastitim zahtjevima kako bi optimizirali fragmente DNA potrebne veličine.
A-2
a) Vrijeme fragmentacije nije optimizirano Ako je fragmentirana DNA premala ili prevelika, molimo vas da pogledate Smjernice za odabir vremena fragmentacije navedene u uputama za određivanje vremena reakcije i koristite ovu vremensku točku kao kontrolu, dodatno postavite reakcijski sustav za produljenje ili skraćivanje 3 minute radi preciznije prilagodbe vremena fragmentacije.
A-3
Abnormalna raspodjela DNK po veličini nakon tretmana fragmentacije
a) Neispravna metoda odmrzavanja reagensa za fragmentaciju ili se reagens nakon odmrzavanja nije potpuno pomiješao. Odmrznite 5 × reagens za miješanje fragmentacijske enzima na ledu. Nakon odmrzavanja ravnomjerno promiješajte reagens laganim trzanjem po dnu epruvete. Ne vrtložite reagens!
b) Ulazni uzorak DNA sadrži EDTA ili druge zagađivače. Osiromašenje iona soli i kelatnih sredstava u koraku pročišćavanja DNA osobito je važno za uspjeh eksperimenta. Ako se DNK otopi u 1 × TE, upotrijebite metodu navedenu u uputama za izvođenje fragmentacije. Ako je koncentracija EDTA u otopini nesigurna, preporučuje se pročišćavanje DNA i otapanje u deioniziranoj vodi radi kasnije reakcije.
c) Netočna početna kvantifikacija DNK Veličina fragmentirane DNA usko je povezana s količinom unete DNK. Prije tretmana fragmentacije, točna kvantifikacija DNA pomoću Qubita, Picogreena i drugih metoda bitna je za određivanje točne količine DNA u reakcijskom sustavu.
d) Priprema reakcijskog sustava ne slijedi upute Priprema fragmentiranog reakcijskog sustava mora se provoditi na ledu strogo prema uputama. Kako bi se osigurao najbolji učinak, sve reakcijske komponente treba staviti na led i pripremiti reakcijski sustav nakon potpunog hlađenja. Nakon što je priprema završena, prelistajte ili pipetom dobro promiješajte. Nemojte vrtjeti!
1. Nepravilna metoda miješanja (vrtlog, snažne oscilacije itd.) Uzrokovat će abnormalnu raspodjelu fragmenata knjižnice (kao što je prikazano na sljedećoj slici), što će utjecati na kvalitetu knjižnice. Stoga, prilikom pripreme reakcijske otopine za fragmentacijsku mješavinu, nježno pipetirajte gore -dolje za miješanje ili upotrijebite vršak prsta za jednolično trzanje i miješanje. Pazite da se ne pomiješate s vrtlogom.

2. Za izgradnju knjižnice mora se koristiti DNK visoke čistoće
■ Dobar integritet DNA: Traka elektroforeze je veća od 30 kb, bez praćenja
■ OD260/230:> 1.5
■ OD260/280: 1,7-1,9
3. Količina ulazne DNK mora biti točna Predlaže se korištenje Qubit i PicoGreen metoda za kvantificiranje DNA, umjesto Nanodropa.
4. Mora se odrediti sadržaj EDTA u otopini DNA. EDTA ima veliki utjecaj na reakciju fragmentacije. Ako je sadržaj EDTA visok, potrebno je provesti pročišćavanje DNA prije sljedećeg testa.
5. Reakcijska otopina za fragmentaciju mora se pripremiti na ledu. Proces fragmentacije osjetljiv je na temperaturu i vrijeme reakcije (osobito nakon dodavanja pojačivača). Kako biste osigurali točnost vremena reakcije, pripremite reakcijski sustav na ledu.
6. Vrijeme reakcije fragmentacije mora biti točno Vrijeme reakcije u koraku fragmentacije izravno će utjecati na veličinu produkata fragmenata, čime će utjecati na raspodjelu veličine fragmenata DNA u biblioteci.
1. Koja vrsta uzorka je primjenjiva na ovaj komplet?
Primjenjivi tip uzorka ovog kompleta može biti ukupna RNA ili pročišćena mRNA s dobrim integritetom RNK. Ako se za izgradnju knjižnice koristi ukupna RNA, preporučuje se prvo upotrijebiti komplet za iscrpljivanje rRNA (Cat#4992363/4992364/4992391) za uklanjanje rRNA.
2. Mogu li se uzorci FFPE -a koristiti za izradu biblioteke s ovim kompletom?
MRNA u uzorcima FFPE -a bit će degradirana u određenoj mjeri, uz relativno loš integritet. Kada se ovaj komplet koristi za izgradnju knjižnice, preporuča se optimizirati vrijeme fragmentacije (skratiti vrijeme fragmentacije ili ne izvršiti fragmentaciju).
3. Koristeći korak odabira veličine koji je naveden u priručniku za proizvod, što bi moglo uzrokovati da umetnuti segment izgleda blago odstupanje?
Odabir veličine provodi se u skladu sa korakom odabira veličine u ovom priručniku za proizvod. Ako postoji odstupanje, razlog bi mogao biti u tome što magnetske kuglice nisu uravnotežene na sobnoj temperaturi ili nisu potpuno pomiješane, pipeta nije točna ili je tekućina ostala u vrhu. Za eksperiment se preporučuje korištenje vrhova s niskom adsorpcijom.
4. Izbor adaptera u izgradnji knjižnice
Komplet za izgradnju knjižnice ne sadrži adapterski reagens, pa se preporučuje korištenje ovog kompleta zajedno s TIANSeq-ovim adapterom s jednim indeksom (Illumina) (4992641/4992642/4992378).
5. QC knjižnice
Kvantitativno otkrivanje knjižnice: Qubit i qPCR se koriste za određivanje masene koncentracije i molarne koncentracije biblioteke. Rad je strogo u skladu s priručnikom za proizvod. Koncentracija knjižnice općenito će zadovoljiti zahtjeve NGS sekvenciranja. Otkrivanje raspona distribucije knjižnica: Korištenje Agilent 2100 Bioanalyzera za otkrivanje raspona distribucije knjižnice.
6. Odabir broja ciklusa pojačanja
Prema uputama, broj ciklusa PCR-a je 6-12, a broj potrebnih ciklusa PCR-a potrebno je odabrati prema unosu uzorka. U knjižnicama visokog prinosa obično dolazi do prekomjernog pojačanja u različitim stupnjevima, što se očituje nešto većim vrhom nakon vrha ciljnog raspona u otkrivanju bioanalizatora Agilent 2100, ili je otkrivena koncentracija Qubita niža od one u qPCR-u. Blago pojačanje normalna je pojava koja ne utječe na redoslijed knjižnica i kasniju analizu podataka.
7. Šiljci se pojavljuju u profilu detekcije bioanalizatora Agilent 2100
Pojava šiljaka u detekciji bioanalizatora Agilent 2100 posljedica je neravnomjerne fragmentacije uzoraka, gdje će biti više fragmenata određene veličine, a to će postati vidljivije nakon obogaćivanja PCR -om. U ovom slučaju, predlaže se da se ne izvrši odabir veličine, odnosno da se uvjet fragmentacije postavi na 94 ° C tijekom 15 minuta inkubacije, gdje je raspodjela fragmenata mala i koncentrirana, a homogenost se može poboljšati.
Kategorije proizvoda
ZAŠTO ODABRATI NAS
Naša tvornica od svog osnutka razvija proizvode prve svjetske klase pridržavajući se načela
prvo kvalitete. Naši su proizvodi stekli izvrsnu reputaciju u industriji i vrijedno povjerenje među novim i starim kupcima.








